Bioquímica Computacional de Receptores de Membrana
La simulación computacional de biomoléculas incluye el modelado tridimensional de receptores, la simulación de sus mecanismos de activación y el esclarecimiento de las bases moleculares de su regulación por otras moléculas. Los métodos de simulación se han afianzado como un componente básico para comprender la bioquímica y farmacología molecular, así como para el diseño de fármacos.
En nuestro laboratorio, nos hemos especializado en desarrollar métodos computacionales para responder a cada uno de estos puntos y aplicar nuestros protocolos para comprender el mecanismo de señalización de los receptores de membrana, con especial atención a los receptores acoplados a proteína G (GPCR), posiblemente la familia más grande de fármacos. Nuestras áreas de interés incluyen i) comprender y mejorar los mecanismos reguladores mediante modulación alostérica, ii) diseño de un espacio químico novedoso con perfiles farmacológicos mejorados, iii) desarrollo de protocolos de modelado altamente eficientes basados en la física tanto para detectar compuestos químicos de interés, como para predecir el efecto de mutaciones patogénicas.
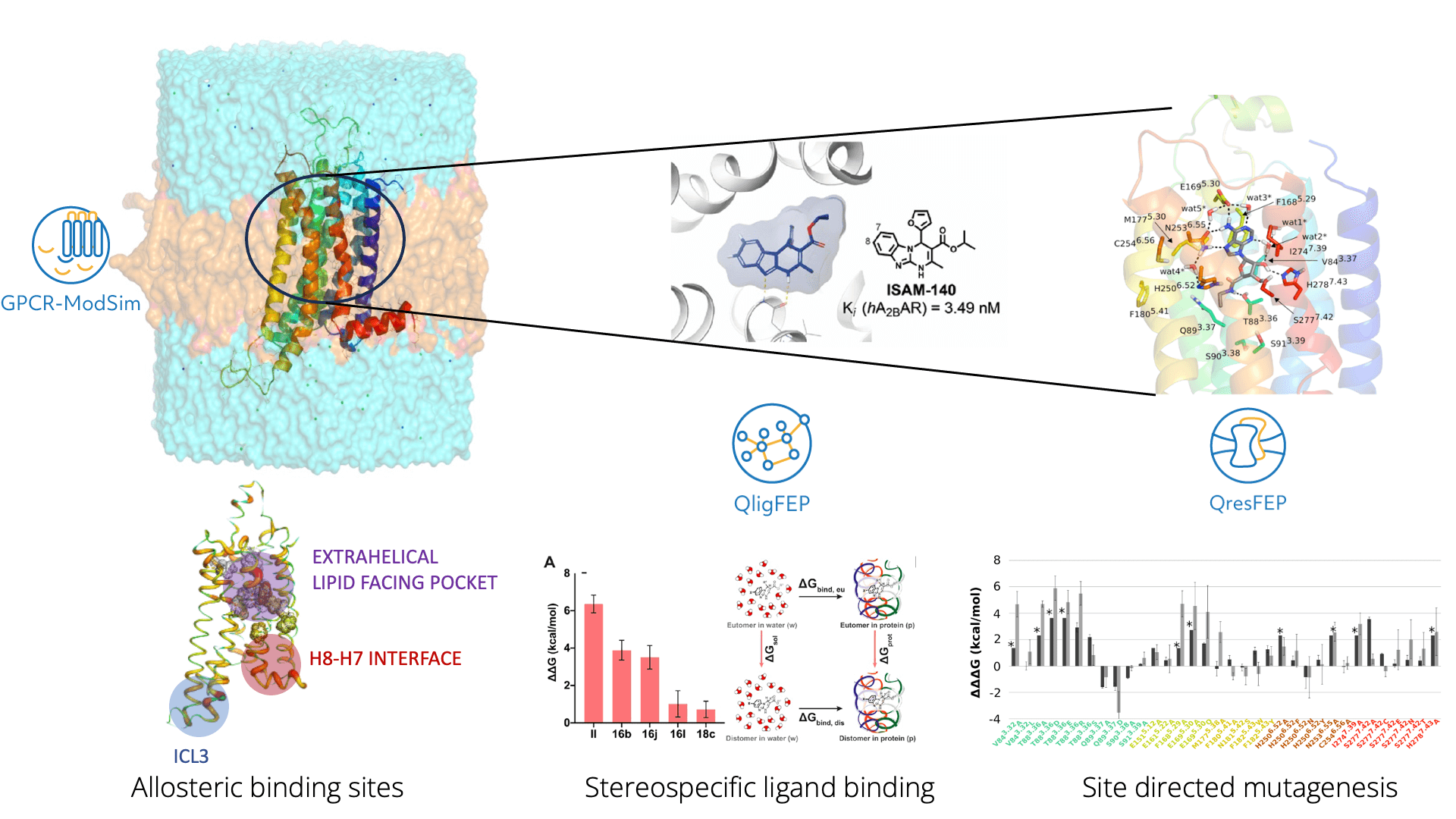
Nuestros métodos de simulación multiescala, nos permiten localizar sitios de unión alostéricos (izquierda, receptor A1 de adenosina), determinar el modo de unión estereoespecífica de una serie de antagonistas del receptor A2B de adenosina, como moduladores inmunológicos en posibles tratamientos de cancer (centro) o determinar el efecto de mutaciones puntuales en la unión a ligando en el receptor A2A de la misma familia (derecha), un método con potenciales aplicaciones en predicción de selectividad o resistencias.

Responsable de laboratorio
Email: h.g.teran@cinn.es
